PR
X
Free Space
〖政治の貧困〗緊急銃猟って、政府、国会の無為無策の結晶か?
〖政治の貧困〗公共事業の誘致が地域政党の生きる道?万博の次は副首都?
〖政治の貧困〗物価高対策はどうなるの?理屈が破綻している?
〖OCNモバイルONE〗そろそろ終了が近いのか?格安SIMサービスの弱点を利用して解約させるのか??非論理的で驚愕の対応!
〖超便利なTailscale VPN〗ロケフリ、アニメロッカーだけでなく、RDPも便利に
【消費税減税】消費税減税に反対の構図とは?
【ソニーロケフリのDDNS終了】ソニーのロケフリのDDNS(ダイナミックDNS)サービス停止後にロケフリを利用する方法
【OCNモバイル】末期に突入なのか?SIM不良って何?
【ミニPC】もうミニパソコンでええやん(いいじゃん)という感じです
【能登半島地震】無法地帯に4G防犯カメラ
〖政治の貧困〗公共事業の誘致が地域政党の生きる道?万博の次は副首都?
〖政治の貧困〗物価高対策はどうなるの?理屈が破綻している?
〖OCNモバイルONE〗そろそろ終了が近いのか?格安SIMサービスの弱点を利用して解約させるのか??非論理的で驚愕の対応!
〖超便利なTailscale VPN〗ロケフリ、アニメロッカーだけでなく、RDPも便利に
【消費税減税】消費税減税に反対の構図とは?
【ソニーロケフリのDDNS終了】ソニーのロケフリのDDNS(ダイナミックDNS)サービス停止後にロケフリを利用する方法
【OCNモバイル】末期に突入なのか?SIM不良って何?
【ミニPC】もうミニパソコンでええやん(いいじゃん)という感じです
【能登半島地震】無法地帯に4G防犯カメラ
テーマ: 医療・健康ニュース(4172)
カテゴリ: データ分析
日々の感染者数のデータがあれば、実効再生産数・Rtの計算ができるWebサイト(shinyアプリのページ)がありました。ベイジアンの手法による計算のようです。
https://shiny.dide.imperial.ac.uk/epiestim/
「R」のパッケージのshinyアプリ版ということのようです。
なお、このアプリは、 「Improved inference of time-varying reproduction numbers during infectious disease outbreaks」 という論文に基づいています。
日々の感染確認者数のデータは、例えば、後述の試作のダッシュボードのグラフの右上のメニューからダウンロードできます。累積のデータではなく、日々の新規数のデータ(Daily New Cases)を利用します。
ダウンロードしたデータの日々のケースのデータの列の部分だけを残して保存し、上記のアプリのサイトにアップロードすれば計算結果とグラフが得られます。
データの列のヘッダーはなしでもありでもどちらでも構いません。読み込む際に指定できるようになっています。
別途、発生 間隔(Serial interval:英文しかないので、日本語の用語がわかりません)の平均値、標準偏差などのパラメータ が必要だったりしますが、過去の伝染病での感染間隔のデータ(例えば2009年の新型インフルエンザの値)を適用することで、とりあえず計算はできます。 → その後、いろいろと調べて、西浦先生らの論文の発症間隔(Serial interval)のパラメータを入力して計算できました。
発症間隔は、1次感染の発症から、2次感染の発症までの期間のようです。つまり、ある人が発症した時点から、その発症した人経由で感染した別の人が発症する時点までの期間のことのようです。
アプリには、発症間隔の平均値の標準偏差だけでなく、標準偏差の平均値や標準偏差などの入力欄があるところはベイズ統計的ですが、今のところ、各入力欄にどのような値を入力すればいいのかよくわかりませんが、95%信頼 区 間(信用区間?)の値を入力するといいのかもしれません。 → その後、いろいろと調べて、西浦先生らの論文の発症間隔のパラメータを入力して計算できました。
このアプリでの計算では、原則として発症間隔についての調査データが必要とされるようです。対策本部の利用しているデータが公開されると、このアプリでより適切な計算ができるようになるのではないでしょうか。
山中先生が別の計算方法で計算されていますが、ある研究論文にあった発症間隔の平均値6.3、標準偏差4.2という値を利用しているようです。
西浦先生らによる論文「Serial interval of novel coronavirus (COVID-19) infections」 というのがあったので、この論文中で言及されている発症間隔の値を用いるのもよさそうです。
この論文では、発症間隔の中央値が4.0ですが、平均値と標準偏差は「 4.7 days (95% CrI: 3.7, 6.0) and 2.9 days (95% CrI: 1.9, 4.9)」ということなので、この平均値と標準偏差の値と、それぞれの95%信頼区間の値をアプリに入力すればよさそうです。でも、「標準偏差の標準偏差」の値のデータがありません。標準偏差の信頼区間から標準偏差の標準偏差を逆算できるのかもしれませんが、ポアソン分布での計算のようなので単純ではなさどうです。とりあえず1としておきます。 → その後、いろいろと調べて、西浦先生らの論文の発症間隔のパラメータを入力して計算できました。
→アプリの途中のメニューで「 Parametric without uncertainty (offset gamma)」を選んで、 「uncertaintyなし」の設定 に すれば、発症間隔の平均値と標準偏差の二つの値を入力するだけで計算できました。平均を4.7
4.8、標準偏差を 2.9
2.3としました。
【5月17日:発病間隔のパラメータを平均4.8、標準偏差2.3に変更します。18日のデータ更新から反映させます。日本科学技術ジャーナリスト会議のニコニコ動画で、利用されているパラメータがわかりました。これまで、同じ論文中の別のパラメータを使用していました】
政府の対策本部の実効再生産数の計算には、この論文中の発生間隔のパラメータが利用されている可能性が高いのではないかと思います。
この西浦先生らの論文には、「潜伏期間よりも感染の間隔が短い場合がある」という内容があり、発症前から感染力がある、という話はこの論文に基づいているようです。
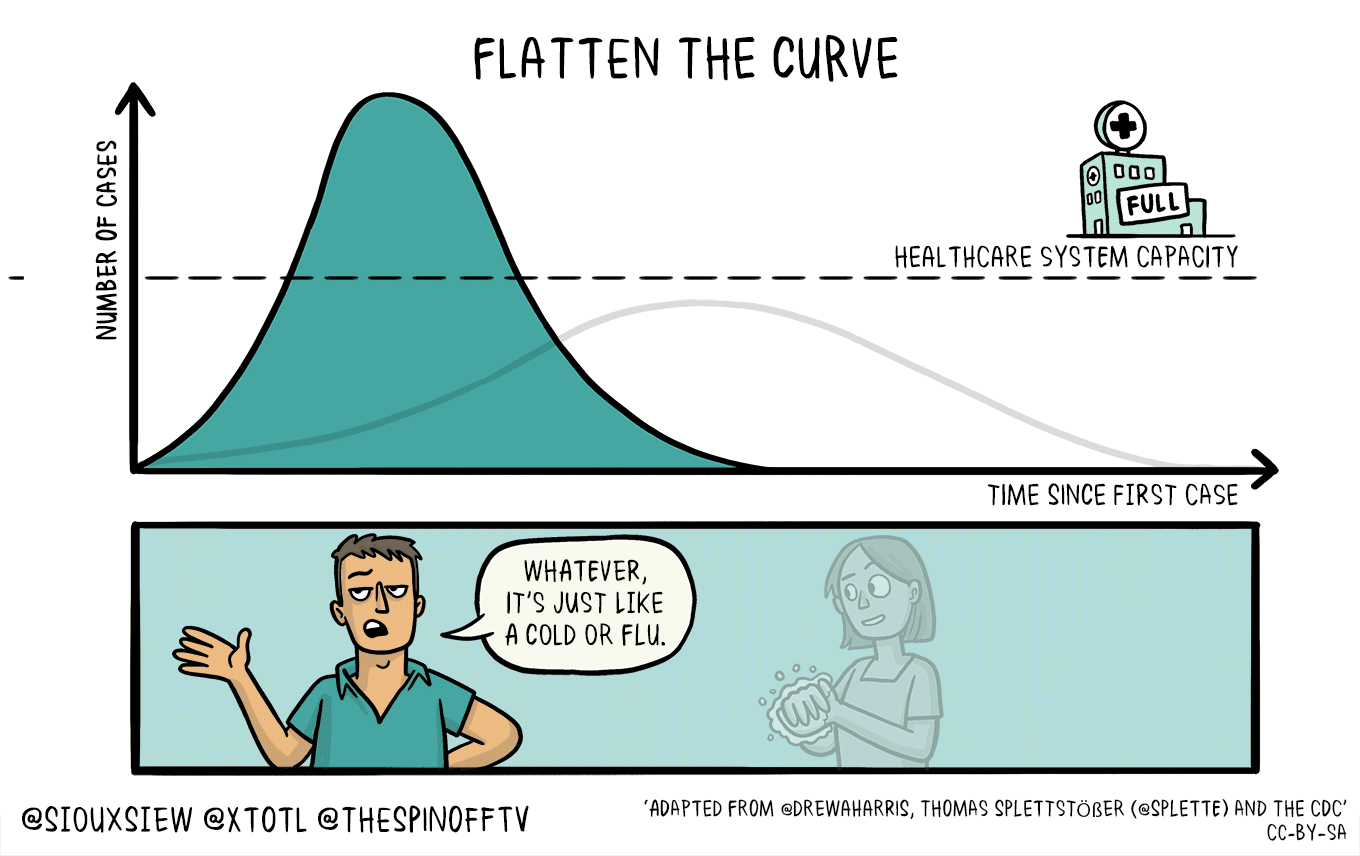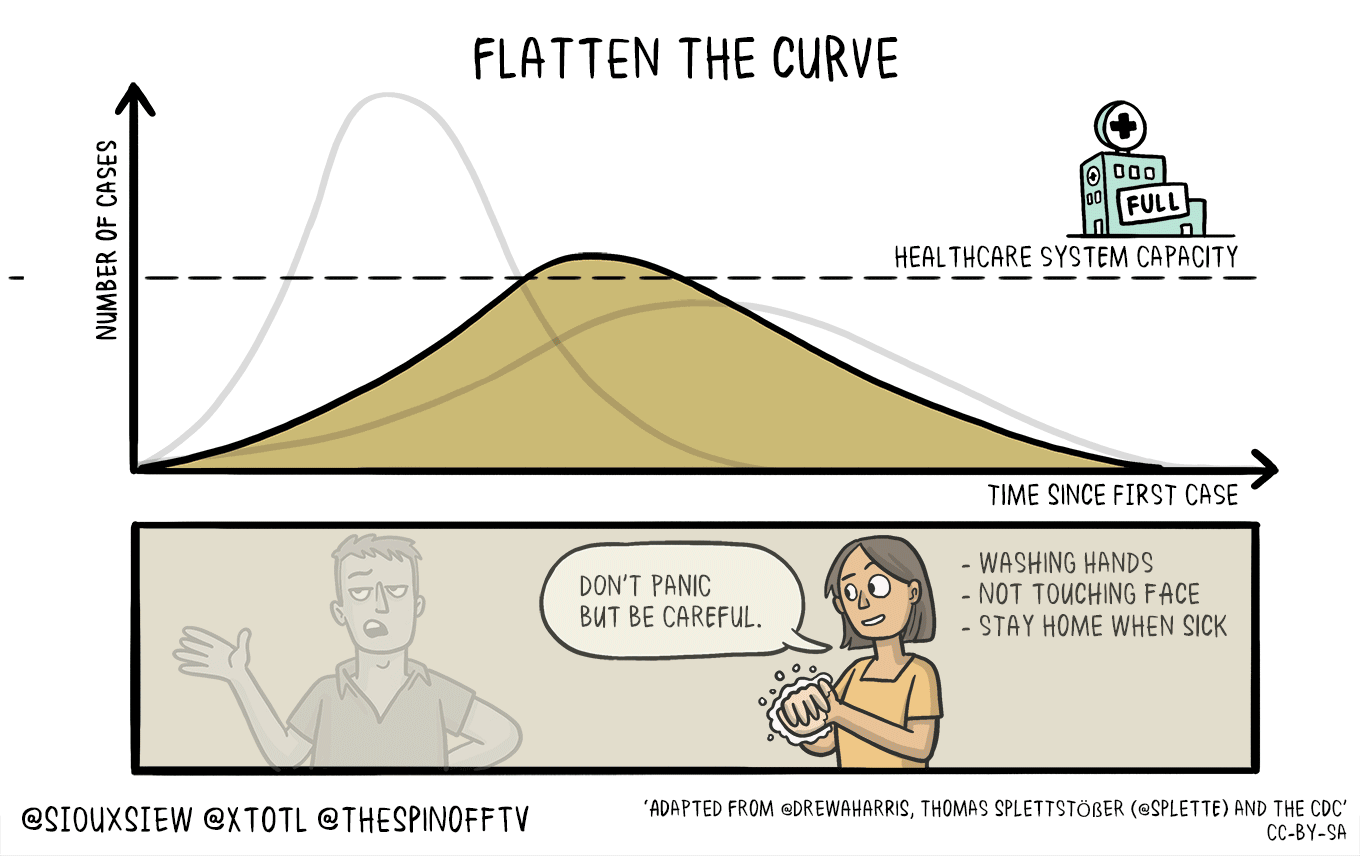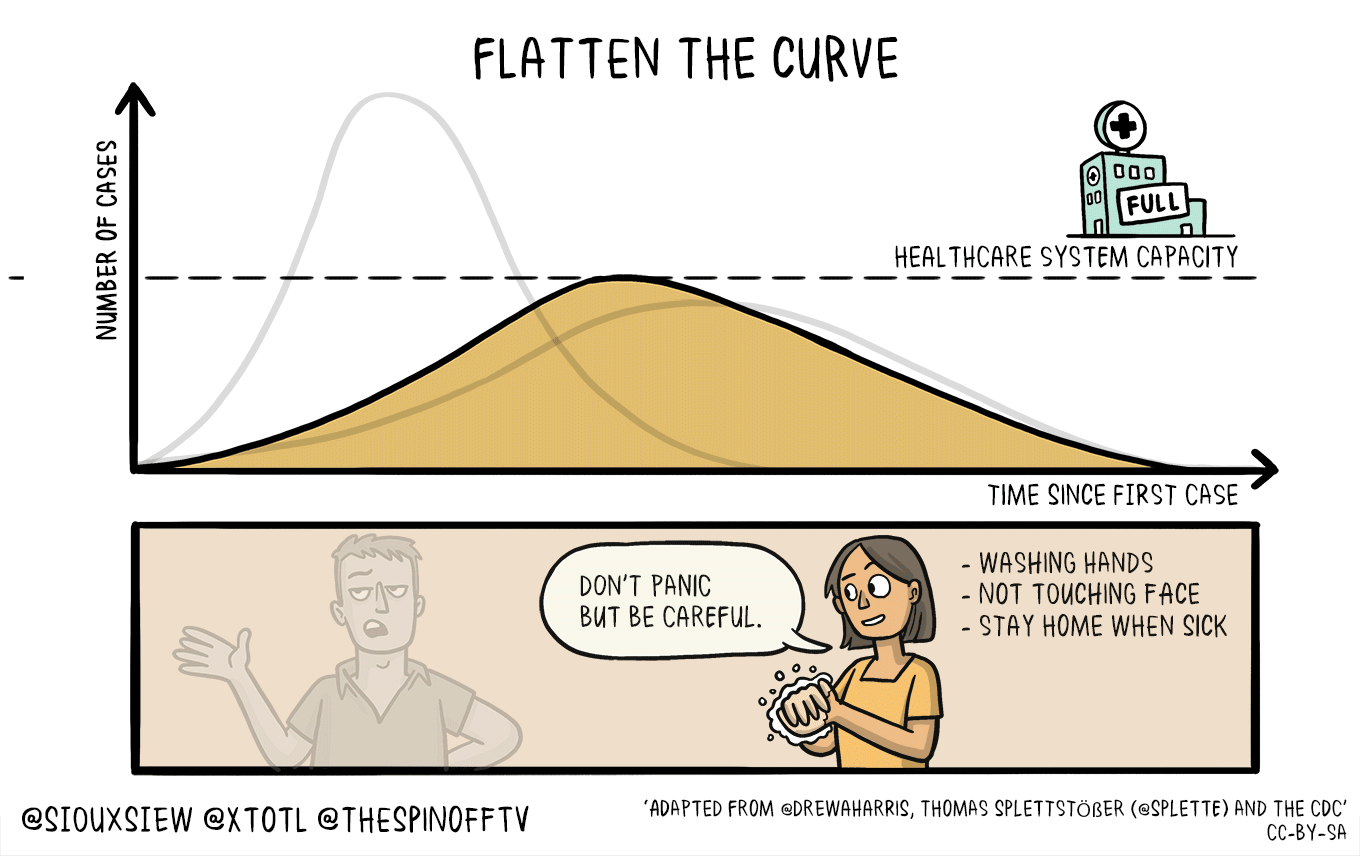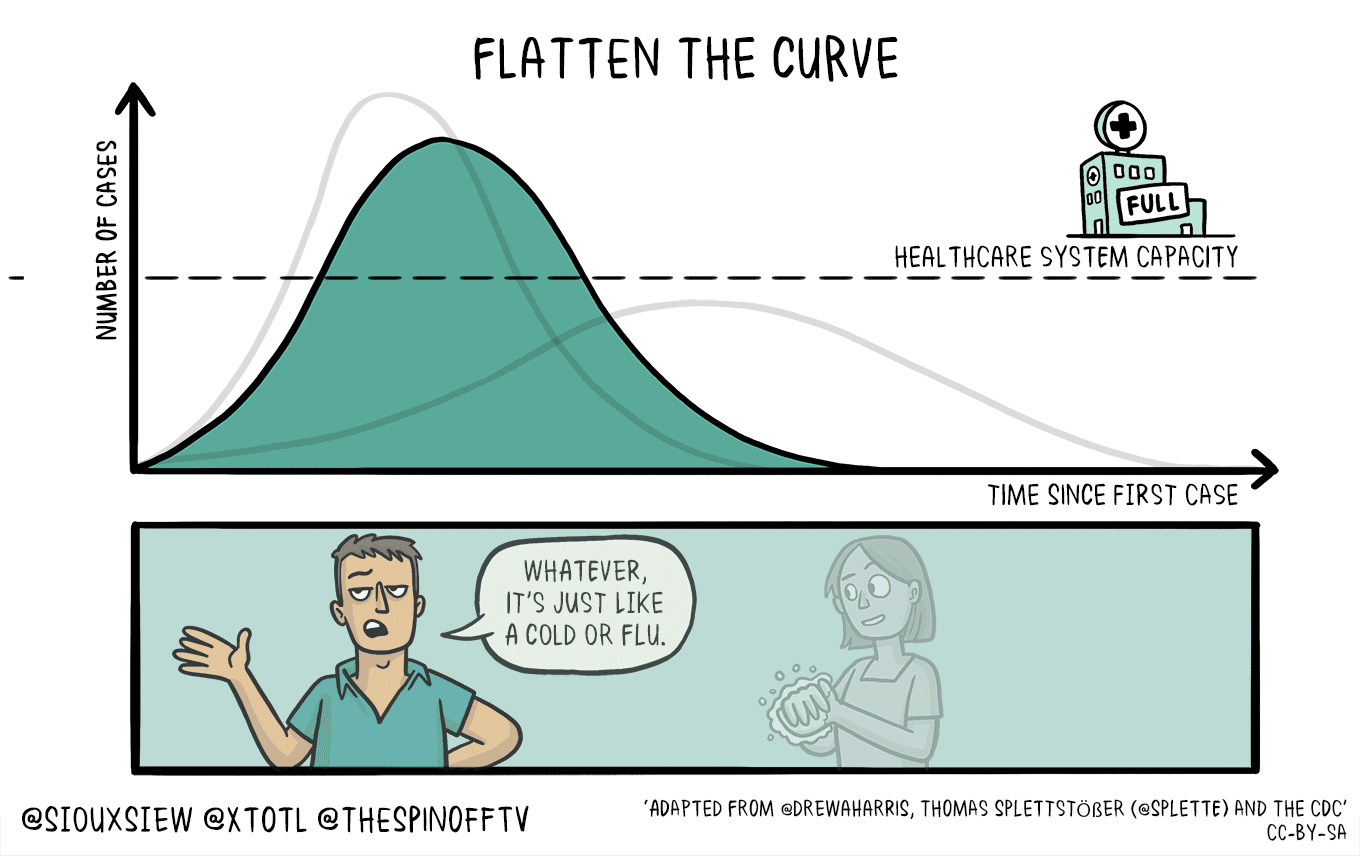
下図は、日本の日々の感染確認者数のデータを、ECDCデータのダッシュボードからダウンロードし、2月以降のデータで計算したものです。
Webアプリによって、下記のグラフとRtの計算結果のデータ表を得ることができます。データ表はcsvファイルでダウンロードできます。
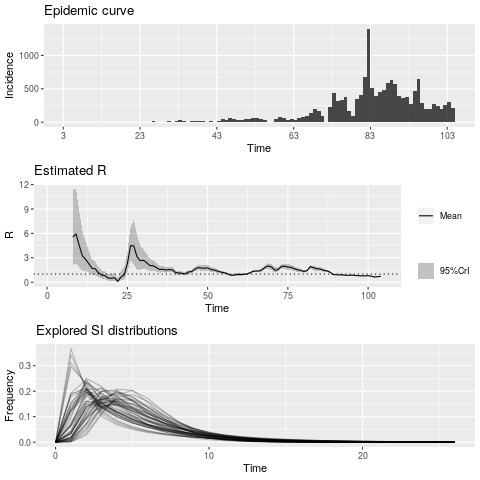
発症間隔を変えて、どのような結果になるのかのシミュレーションにも利用できそうではあります。
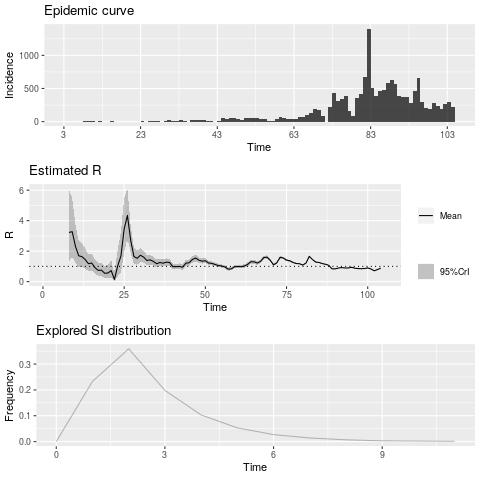
↓「uncertaintyなし」に設定して、 西浦先生らの論文の発症間隔のパラメータの、 平均4.7、標準偏差2.9を利用した場合。最新のRtは、0.74です。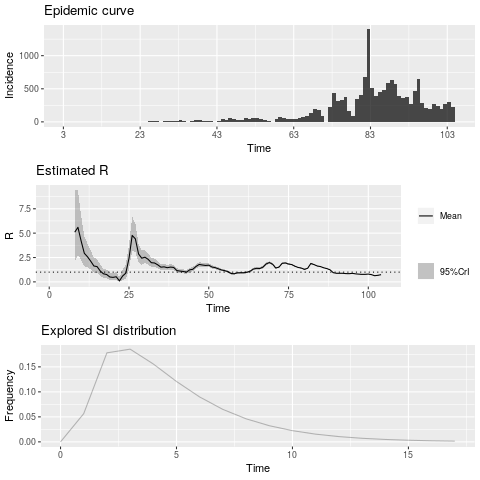
【Rのパッケージによる計算】
「EpiEstim package for R」の コードの記載のあるブログ を参考にして、何とかできました。
https://timchurches.github.io/blog/posts/2020-02-18-analysing-covid-19-2019-ncov-outbreak-data-with-r-part-1/#estimating-changes-in-the-effective-reproduction-number
【5月17日:発病間隔のパラメータを平均4.8、標準偏差2.3に変更します。18日のデータ更新から反映させます。日本科学技術ジャーナリスト会議のニコニコ動画で、利用されているパラメータがわかりました。これまで、同じ論文中の別のパラメータを使用していました】
下記は、コードの例です。
データとしては、ECDCのデータを読み込んで、日本のデータを抽出したものを利用しています。1月以前のデータも含んでいます。
コードで計算できるようになったので、入力データを入れ替えるだけで多くの国や地域での計算が簡単にできるようになりました。
計算結果をダッシュボードでグラフ化できるようにしていきたいと考えています。
【コード例:ECDCのサイトからのデータ読み込みとRtの計算ができます】
df_ECDC <- read.csv("https://opendata.ecdc.europa.eu/covid19/casedistribution/csv", na.strings = "", fileEncoding = "UTF-8-BOM")
df_ECDCJapan <- dplyr::filter(df_ECDC,geoId == "JP")
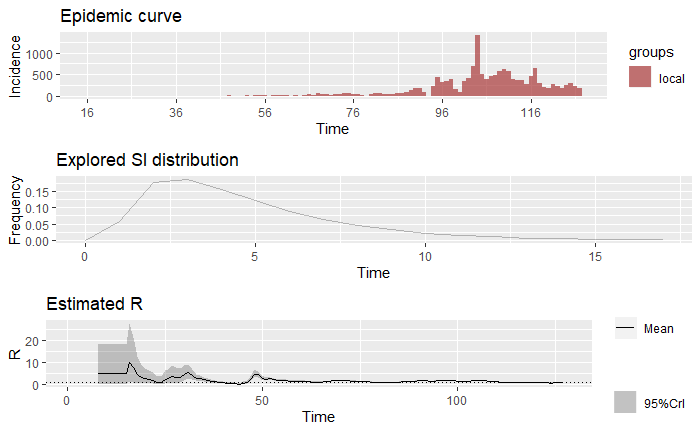
結局、一般的な「実効再生産数・Rt」の計算で使われているデータは、「日々の感染確認者数のデータ」と、「発症間隔(1次感染者の発症から2次感染者の発症までの日数)のパラメータ」のみということなので、日々の感染確認者数の増加や減少を反映している指標に過ぎない、という見方ができるようです。
何か、賢そうな名前の指標ですが、それほど役立つものでもなさそうです。
実効再生産数・Rtには、わかりやすい概念という側面と、計算で推定される指標という側面があり、概念の方から見ると、「実効再生産数」ってすごいと思ってしまいますが、計算による推定方法の仕組みを知ると、期待外れになるかもしれません。
「概念」と「推定される数値の指標」は別物として考えるのがいいのかもしれません。
--------------------------------------------------------------------------

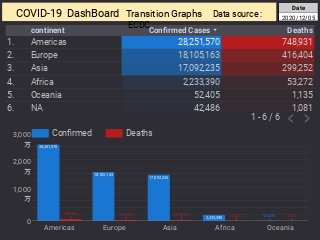
【ダッシュボード 「COVID-19 Transition Graphs」 を試作】
中国本土以外の地域への感染が拡大しているため、国別、地域別の感染者数の推移を簡単に確認できるダッシュボードを試作しています。
随時、ページを追加しています。グラフのデータは、右上部分の操作でダウンロードすることができます。
アメリカの「地域別の変数」を前処理して、「州別」での推移をグラフ化できるようにしました。
また、州コードのフィールドを作成してコロプレス地図も作成しています。
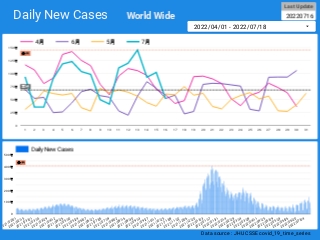
楽天ブログでは「iframe」タグが使えないので、Bloggerのページから利用できるようにしています。
無料で利用できる、グーグルの「データポータル」のダッシュボードです。データさえあれば、簡単に作成できます。「国」別、「地域」別に日ごとの感染者数の推移を見ることができます。
↓ダッシュボードの試作です。下記リンクのページから利用できます。

ジョンズ・ホプキンス大学の 「JHU CSSE」の「Covid19 Daily Reports」のデータを利用しています。
直近のアメリカのデータは地域分類が細かくなっていて、1日当たり2千行くらいになっています。

★おすすめの記事


◆How Windows Sonic looks like.:Windows Sonic for Headphonesの音声と2chステレオ音声の比較:7.1.2chテストトーンの比較で明らかになった違い:一目で違いがわかりました
https://shiny.dide.imperial.ac.uk/epiestim/
「R」のパッケージのshinyアプリ版ということのようです。
なお、このアプリは、 「Improved inference of time-varying reproduction numbers during infectious disease outbreaks」 という論文に基づいています。
日々の感染確認者数のデータは、例えば、後述の試作のダッシュボードのグラフの右上のメニューからダウンロードできます。累積のデータではなく、日々の新規数のデータ(Daily New Cases)を利用します。
ダウンロードしたデータの日々のケースのデータの列の部分だけを残して保存し、上記のアプリのサイトにアップロードすれば計算結果とグラフが得られます。
データの列のヘッダーはなしでもありでもどちらでも構いません。読み込む際に指定できるようになっています。
別途、発生 間隔(Serial interval:英文しかないので、日本語の用語がわかりません)の平均値、標準偏差などのパラメータ が必要だったりしますが、過去の伝染病での感染間隔のデータ(例えば2009年の新型インフルエンザの値)を適用することで、とりあえず計算はできます。 → その後、いろいろと調べて、西浦先生らの論文の発症間隔(Serial interval)のパラメータを入力して計算できました。
発症間隔は、1次感染の発症から、2次感染の発症までの期間のようです。つまり、ある人が発症した時点から、その発症した人経由で感染した別の人が発症する時点までの期間のことのようです。
アプリには、発症間隔の平均値の標準偏差だけでなく、標準偏差の平均値や標準偏差などの入力欄があるところはベイズ統計的ですが、今のところ、各入力欄にどのような値を入力すればいいのかよくわかりませんが、95%信頼 区 間(信用区間?)の値を入力するといいのかもしれません。 → その後、いろいろと調べて、西浦先生らの論文の発症間隔のパラメータを入力して計算できました。
このアプリでの計算では、原則として発症間隔についての調査データが必要とされるようです。対策本部の利用しているデータが公開されると、このアプリでより適切な計算ができるようになるのではないでしょうか。
山中先生が別の計算方法で計算されていますが、ある研究論文にあった発症間隔の平均値6.3、標準偏差4.2という値を利用しているようです。
西浦先生らによる論文「Serial interval of novel coronavirus (COVID-19) infections」 というのがあったので、この論文中で言及されている発症間隔の値を用いるのもよさそうです。
この論文では、発症間隔の中央値が4.0ですが、平均値と標準偏差は「 4.7 days (95% CrI: 3.7, 6.0) and 2.9 days (95% CrI: 1.9, 4.9)」ということなので、この平均値と標準偏差の値と、それぞれの95%信頼区間の値をアプリに入力すればよさそうです。でも、「標準偏差の標準偏差」の値のデータがありません。標準偏差の信頼区間から標準偏差の標準偏差を逆算できるのかもしれませんが、ポアソン分布での計算のようなので単純ではなさどうです。とりあえず1としておきます。 → その後、いろいろと調べて、西浦先生らの論文の発症間隔のパラメータを入力して計算できました。
→アプリの途中のメニューで「 Parametric without uncertainty (offset gamma)」を選んで、 「uncertaintyなし」の設定 に すれば、発症間隔の平均値と標準偏差の二つの値を入力するだけで計算できました。平均を
【5月17日:発病間隔のパラメータを平均4.8、標準偏差2.3に変更します。18日のデータ更新から反映させます。日本科学技術ジャーナリスト会議のニコニコ動画で、利用されているパラメータがわかりました。これまで、同じ論文中の別のパラメータを使用していました】
新規コロナウイルス(COVID-19)感染の連続間隔。西浦H 1、リントンNM 2、アフメチャノフAR 2。
概要目的:28の感染者と感染者のペアに関する情報から、新規コロナウイルス(COVID-19)のシリアル間隔を推定します。方法:公開された研究記事とケース調査レポートから、一次症例(感染者)と二次症例(感染者)の発症日を収集しました。データの信頼性を主観的にランク付けし、完全なデータセット(n = 28)とレポートで最も確実性の高いペアのサブセット(n = 18)の両方で分析を行いました。さらに、流行がまだ成長段階にあるため、データの正しい切り捨てを調整します。結果:正しい切り捨てを考慮してすべてのペアを分析し、シリアル間隔の中央値を4.0日と推定しました(95%信頼できる間隔[CrI]:3.1、4.9)。データを最も特定のペアのみに制限すると、シリアル間隔の中央値は4.6日と推定されました(95%CrI:3.5、5.9)。結論:COVID-19のシリアル間隔は、その潜伏期間の中央値に近いか、それよりも短いです。これは、二次感染のかなりの部分が発症前に発生する可能性があることを示唆しています。COVID-19のシリアルインターバルは、重症急性呼吸器症候群(SARS)のシリアルインターバルよりも短く、SARSシリアルインターバルを使用して行われた計算でバイアスが生じる可能性があることを示しています。
ResultsWe were able to obtain data on 28 infector-infectee pairs(see Supplementary Table). Of these, 12 pairs were familyclusters. Accounting for right truncation and analyzing allpairs, the model using the lognormal distribution was selectedas the best-fit model (WAIC = 224.0), while no significantdifferences from other models were identified. The medianserial interval was estimated at 4.0 days (95% credible interval[CrI]: 3.1, 4.9) while the mean and standard deviation (SD) ofthe serial interval were estimated at 4.7 days (95% CrI: 3.7, 6.0)and 2.9 days (95% CrI: 1.9, 4.9), respectively. Withouttruncation, the model using the lognormal distribution wasalso the best-fit model (WAIC = 128.0) with the median serialinterval estimated at 3.9 days (95% CrI: 3.1, 4.8).
政府の対策本部の実効再生産数の計算には、この論文中の発生間隔のパラメータが利用されている可能性が高いのではないかと思います。
この西浦先生らの論文には、「潜伏期間よりも感染の間隔が短い場合がある」という内容があり、発症前から感染力がある、という話はこの論文に基づいているようです。
下図は、日本の日々の感染確認者数のデータを、ECDCデータのダッシュボードからダウンロードし、2月以降のデータで計算したものです。
Webアプリによって、下記のグラフとRtの計算結果のデータ表を得ることができます。データ表はcsvファイルでダウンロードできます。
発症間隔を変えて、どのような結果になるのかのシミュレーションにも利用できそうではあります。

↓
「uncertaintyあり」に設定し、
西浦先生らの論文の発症間隔のパラメータを用いた場合。発症間隔の分布がMCMCで推定されているようです。

↓「uncertaintyなし」に設定して、 西浦先生らの論文の発症間隔のパラメータの、 平均4.7、標準偏差2.9を利用した場合。最新のRtは、0.74です。

【Rのパッケージによる計算】
「EpiEstim package for R」の コードの記載のあるブログ を参考にして、何とかできました。
https://timchurches.github.io/blog/posts/2020-02-18-analysing-covid-19-2019-ncov-outbreak-data-with-r-part-1/#estimating-changes-in-the-effective-reproduction-number
【5月17日:発病間隔のパラメータを平均4.8、標準偏差2.3に変更します。18日のデータ更新から反映させます。日本科学技術ジャーナリスト会議のニコニコ動画で、利用されているパラメータがわかりました。これまで、同じ論文中の別のパラメータを使用していました】
下記は、コードの例です。
データとしては、ECDCのデータを読み込んで、日本のデータを抽出したものを利用しています。1月以前のデータも含んでいます。
コードで計算できるようになったので、入力データを入れ替えるだけで多くの国や地域での計算が簡単にできるようになりました。
計算結果をダッシュボードでグラフ化できるようにしていきたいと考えています。
【コード例:ECDCのサイトからのデータ読み込みとRtの計算ができます】
df_ECDC <- read.csv("https://opendata.ecdc.europa.eu/covid19/casedistribution/csv", na.strings = "", fileEncoding = "UTF-8-BOM")
df_ECDCJapan <- dplyr::filter(df_ECDC,geoId == "JP")
install.packages("EpiEstim")
library(EpiEstim)
plot_Ri(japan_parametric_si)
plot_Ri <- function(estimate_R_obj) {
p_I <- plot(estimate_R_obj, "incid", add_imported_cases = TRUE) # plots the incidence
p_SI <- plot(estimate_R_obj, "SI") # plots the serial interval distribution
p_Ri <- plot(estimate_R_obj, "R")
return(gridExtra::grid.arrange(p_I, p_SI, p_Ri, ncol = 1))
}
japan_parametric_si <- estimate_R(df_ECDCJapan$cases,
method = "parametric_si", config = make_config(list(mean_si = 4.8
,
std_si = 2.3
)))
plot_Ri(japan_parametric_si)
options(max.print=10000)
japan_parametric_si

結局、一般的な「実効再生産数・Rt」の計算で使われているデータは、「日々の感染確認者数のデータ」と、「発症間隔(1次感染者の発症から2次感染者の発症までの日数)のパラメータ」のみということなので、日々の感染確認者数の増加や減少を反映している指標に過ぎない、という見方ができるようです。
何か、賢そうな名前の指標ですが、それほど役立つものでもなさそうです。
実効再生産数・Rtには、わかりやすい概念という側面と、計算で推定される指標という側面があり、概念の方から見ると、「実効再生産数」ってすごいと思ってしまいますが、計算による推定方法の仕組みを知ると、期待外れになるかもしれません。
「概念」と「推定される数値の指標」は別物として考えるのがいいのかもしれません。

--------------------------------------------------------------------------

洗って、繰り返し使える布マスクです。
![[商品価格に関しましては、リンクが作成された時点と現時点で情報が変更されている場合がございます。] [商品価格に関しましては、リンクが作成された時点と現時点で情報が変更されている場合がございます。]](https://hbb.afl.rakuten.co.jp/hgb/1ae15073.43ba9030.1ae15074.eb6248a2/?me_id=1222042&item_id=10024456&m=https%3A%2F%2Fthumbnail.image.rakuten.co.jp%2F%400_mall%2Fandit%2Fcabinet%2Fitem%2Fmask%2Fand-h202018a.jpg%3F_ex%3D80x80&pc=https%3A%2F%2Fthumbnail.image.rakuten.co.jp%2F%400_mall%2Fandit%2Fcabinet%2Fitem%2Fmask%2Fand-h202018a.jpg%3F_ex%3D240x240&s=240x240&t=picttext)
|
長さ調節可フィルターも入る2重構造!洗濯可立体仕様のケアマスク●先行受注●【メール便で送料無料】【メール便A】マスク 布マスク 男女兼用 日本製 国内生産 二重構造 フィルター効果 ユニセックス 洗える 洗濯可能 清潔 花粉症 保湿 コットン 綿
|
【ダッシュボード 「COVID-19 Transition Graphs」 を試作】
中国本土以外の地域への感染が拡大しているため、国別、地域別の感染者数の推移を簡単に確認できるダッシュボードを試作しています。
随時、ページを追加しています。グラフのデータは、右上部分の操作でダウンロードすることができます。
アメリカの「地域別の変数」を前処理して、「州別」での推移をグラフ化できるようにしました。
また、州コードのフィールドを作成してコロプレス地図も作成しています。
楽天ブログでは「iframe」タグが使えないので、Bloggerのページから利用できるようにしています。
無料で利用できる、グーグルの「データポータル」のダッシュボードです。データさえあれば、簡単に作成できます。「国」別、「地域」別に日ごとの感染者数の推移を見ることができます。
↓ダッシュボードの試作です。下記リンクのページから利用できます。

ジョンズ・ホプキンス大学の 「JHU CSSE」の「Covid19 Daily Reports」のデータを利用しています。
直近のアメリカのデータは地域分類が細かくなっていて、1日当たり2千行くらいになっています。
EdgeブラウザやIEブラウザなど、Chromeブラウザ以外での利用の場合はうまく表示されないことがあるようです。
↓ 上記のダッシュボードのデータの出所のサイトです。マップがメインのダッシュボードです

↓WHOのサイトでも、感染者数、地域などの「Situation Report」が日々更新されています。関心がある場合は、一日に一度見るといいのではないかと思います。

--------------------------------------------------------------------------

--------------------------------------------------------------------------

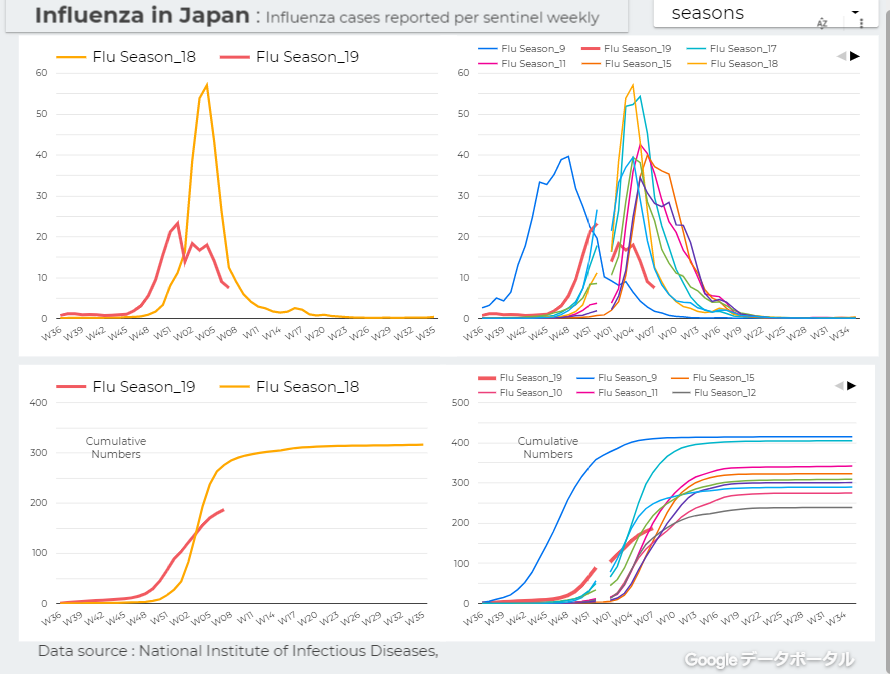
↓日本のインフルエンザの「定点当たり報告数」をグラフ化できるダッシュボードを試作。都道府県別にグラフ化可能です。
------------------------------------------------------
新型コロナウイルス(2019-novel coronavirus)対策もインフルエンザ対策と同じで、手洗い、うがい、マスク着用(咳エチケット)、免疫力アップなどが対策になるようです。
↓ 上記のダッシュボードのデータの出所のサイトです。マップがメインのダッシュボードです

 がんばれ、イタリア。
がんばれ、イタリア。
[イタリア お土産]バッチ チョコレート 12粒入り (イタリア お土産 イタリア 土産 イタリア おみやげ イタリア みやげ)
価格:1448円(税別、送料別) (2020/3/12時点)

楽天で購入
↓WHOのサイトでも、感染者数、地域などの「Situation Report」が日々更新されています。関心がある場合は、一日に一度見るといいのではないかと思います。

--------------------------------------------------------------------------

--------------------------------------------------------------------------

↓日本のインフルエンザの「定点当たり報告数」をグラフ化できるダッシュボードを試作。都道府県別にグラフ化可能です。

------------------------------------------------------
【3/6再入荷】【お一人様3個まで】LEC レック 除菌の【激落ちくん】320ml 除菌 激落ち キッチン アルカリ電解水 配合 アルコールスプレー 除菌スプレー【RCP】【S-659】【キャッシュレス 還元 対象店】
価格:260円(税別、送料別) (2020/3/7時点)

楽天で購入
★おすすめの記事




◆How Windows Sonic looks like.:Windows Sonic for Headphonesの音声と2chステレオ音声の比較:7.1.2chテストトーンの比較で明らかになった違い:一目で違いがわかりました

お気に入りの記事を「いいね!」で応援しよう
[データ分析] カテゴリの最新記事
-
CHATGPTって何しているの? 2023.11.22
-
コスタリカの話題はあまり見られない:ツ… 2022.12.03
-
【新型コロナ】Qatarでは感染確認者数が減… 2022.11.29
【毎日開催】
15記事にいいね!で1ポイント
10秒滞在
いいね!
--
/
--
© Rakuten Group, Inc.



![[商品価格に関しましては、リンクが作成された時点と現時点で情報が変更されている場合がございます。] [商品価格に関しましては、リンクが作成された時点と現時点で情報が変更されている場合がございます。]](https://hbb.afl.rakuten.co.jp/hgb/1ac0e17f.509c33e8.1ac0e180.04cbdc49/?me_id=1219890&item_id=10001366&m=https%3A%2F%2Fthumbnail.image.rakuten.co.jp%2F%400_mall%2Farigatou-nuts%2Fcabinet%2Fpickup%2Fimgrc0091437697.jpg%3F_ex%3D80x80&pc=https%3A%2F%2Fthumbnail.image.rakuten.co.jp%2F%400_mall%2Farigatou-nuts%2Fcabinet%2Fpickup%2Fimgrc0091437697.jpg%3F_ex%3D240x240&s=240x240&t=picttext)
![[商品価格に関しましては、リンクが作成された時点と現時点で情報が変更されている場合がございます。] [商品価格に関しましては、リンクが作成された時点と現時点で情報が変更されている場合がございます。]](https://hbb.afl.rakuten.co.jp/hgb/1aae7a85.f4143c83.1aae7a86.7dc201df/?me_id=1210867&item_id=10054198&m=https%3A%2F%2Fthumbnail.image.rakuten.co.jp%2F%400_gold%2Fgoodlifeshop%2Fgcabi%2Fbct%2F4903320575981.gif%3F_ex%3D80x80&pc=https%3A%2F%2Fthumbnail.image.rakuten.co.jp%2F%400_gold%2Fgoodlifeshop%2Fgcabi%2Fbct%2F4903320575981.gif%3F_ex%3D240x240&s=240x240&t=picttext)






